Alla fine del 2018, in occasione di una conferenza e di un concorso di biologia noto come Critical Assessment of Structure Prediction, la filiale londinese di Alphabet, DeepMind, con sede a Londra, ha dato un’incredibile dimostrazione della sua capacità di dedurre la struttura fisica delle proteine sulla base del loro codice genetico. L’IA di DeepMind, AlphaFold, ha vinto il concorso, facendo le migliori previsioni in 43 dei 90 test. La società non aveva mai partecipato alla conferenza prima d’ora. Anche se impressionante, la tecnologia non era ancora in grado di sostituire i costosi e lunghi metodi sperimentali esistenti per determinare l’aspetto di queste proteine. Tuttavia, il suo ultimo software ci si avvicina.
A novembre, AlphaFold ha nuovamente superato tutti gli altri gruppi concorrenti al CASP.
La tecnologia ha risolto le strutture proteiche su cui altri laboratori stavano lavorando da anni. Gli scienziati pensano che la tecnologia potrebbe avere immense implicazioni per il modo in cui le proteine vengono studiate.
DeepMind è ancora in fase di convalida della sua ultima tecnologia. Ma sta già lavorando con partner accademici e industriali per capire come la tecnologia possa avere il massimo impatto. Un’area ovvia potrebbe essere la scoperta dei farmaci. Comprendere il modo in cui le proteine si piegano in forme tridimensionali è fondamentale per capire come progettare i farmaci. Gli scienziati devono sapere come le varie molecole si agganciano alle proteine e come cambiano il modo in cui operano nell’organismo, per proporre potenziali farmaci.
Ora DeepMind sta convalidando la sua tecnologia e scrivendo un articolo su come funziona il tutto.
In una conversazione con Fast Company, il suo principale e capo del team Pushmeet Kohli spiega come la tecnologia sia stata snellita per fare previsioni migliori sulla struttura tridimensionale della proteina e su cosa significhi per il futuro della sanità.

L’intervista a Pushmeet Kohli, CEO di DeepMind
Come è cambiata la tecnologia AlphaFold rispetto all’anno scorso?
“Nel lavoro precedente, la rete neurale otteneva la sequenza di una proteina e prevedeva quali particolari amminoacidi sarebbero stati vicini l’uno all’altro.
Quindi si trattava di prevedere questo istogramma o questa matrice a distanza, che essenzialmente diceva che questo amminoacido sarebbe stato vicino a quest’altro amminoacido e così via. Poi un secondo modulo di fatto usava quell’informazione per strutturare quell’informazione in 3D.
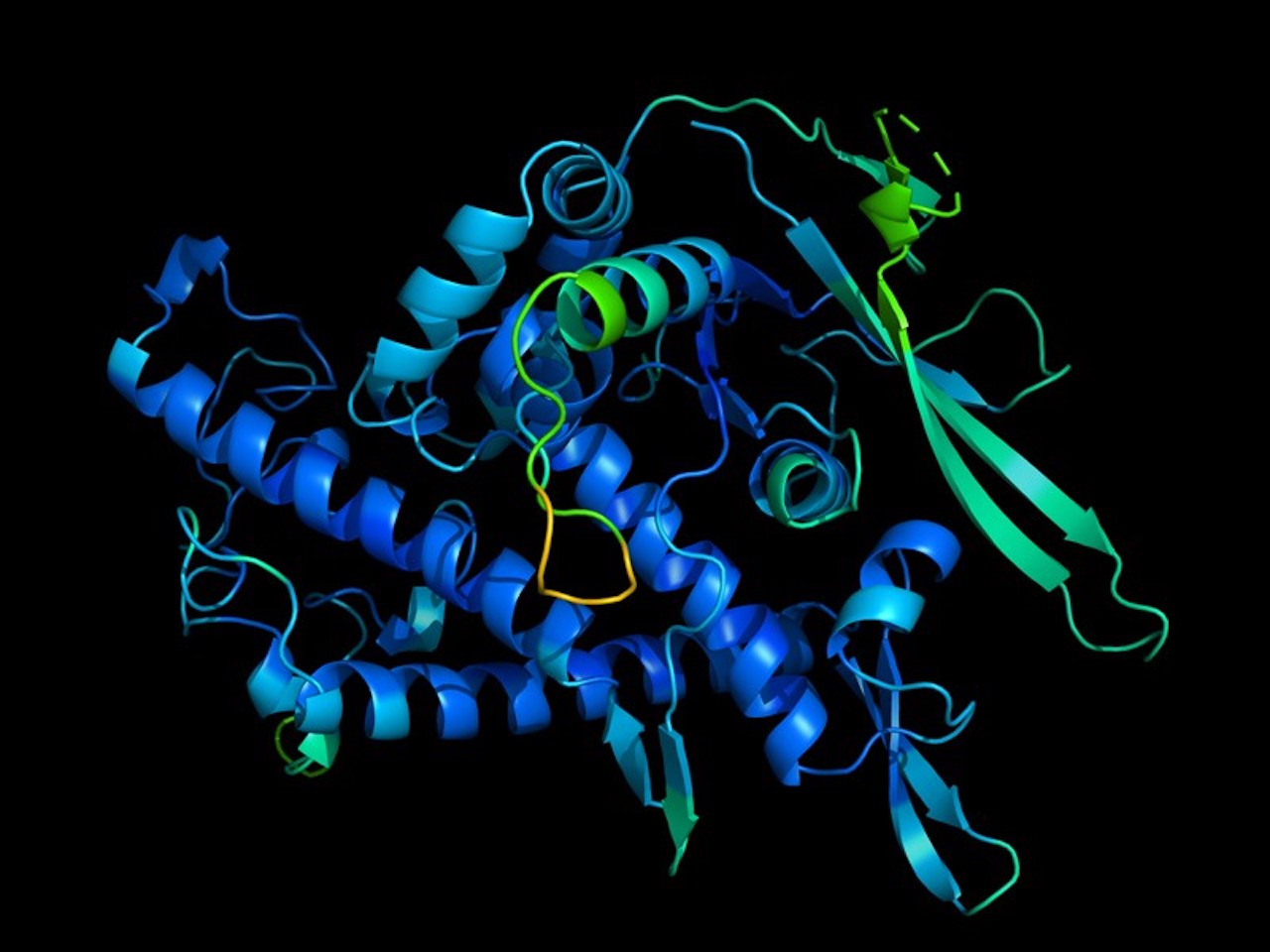
Nel nuovo sistema, questo processo in due fasi non avviene più. Si tratta di una sola rete neurale, che prende solo la sequenza e l’allineamento e fa direttamente una previsione sulla struttura della proteina. Quella rete neurale è modulata con la capacità di generalizzare. A scuola, impariamo il concetto di addizione. Non memorizziamo che tre più quattro è uguale a sette o 23 più 17 è uguale a 40. Comprendiamo il concetto di cosa sia l’addizione e come si possano aggiungere due numeri qualsiasi.
Questa comprensione concettuale è ciò che abbiamo cercato di fare nella rete neurale in modo che non memorizzi che “a questa sequenza corrisponde questa struttura”. Cerca di capire quali concetti sono in gioco, in modo da poter prevedere qualsiasi proteina, non solo le proteine che sono state intuite nei dati di allenamento.
Quali proteine non può prevedere?
Oggi basa la sua comprensione sulla storia evolutiva. Quindi, fondamentalmente, guarda tutte le proteine conosciute e vede come alcuni residui interagiscono. Sta usando queste informazioni per fare previsioni sulla struttura. Ora, se si ha una proteina completamente nuova, che non ha una storia evolutiva – una proteina sintetica, che è stata appena progettata – allora il nostro sistema non sarebbe così preciso come lo sarebbe per le proteine esistenti. Ma se si ha una proteina che è presente in natura, che ha una storia evolutiva, allora il nostro sistema sarà in grado di funzionare abbastanza bene. Nella competizione CASP, siamo stati in grado di vedere che per una grande maggioranza delle proteine in competizione il nostro sistema è stato in grado di produrre previsioni estremamente accurate che sono quasi alla pari con i metodi sperimentali.
Come può questa tecnologia aiutare i ricercatori o i biologi dell’industria?
Diciamo che state cercando di capire i meccanismi di una malattia. Anche prendendo l’esempio della SARS COV-2 [il virus che causa il COVID-19], uno degli elementi più importanti è stato quello di sequenziare prima il virus e poi di capire le sue strutture tridimensionali, dopo di che siamo stati in grado di capire come il virus si stava interfacciando con le cellule del corpo umano. Con questa tecnologia, saremmo stati in grado di accelerare questo processo. Gli sperimentatori sarebbero ora in grado di fare un lavoro migliore e di essere più veloci nel progettare questo esperimento, piuttosto che dover aspettare un paio d’anni mentre qualcuno si occupa della struttura tridimensionale delle proteine.
La velocità è un grosso problema in particolare nella scoperta dei farmaci. Gli esperimenti clinici richiedono molto tempo e spesso falliscono. Questa tecnologia potrebbe aiutare le percentuali di successo?
Assolutamente sì. Trovare la struttura di una proteina richiede molto tempo. I progettatori di farmaci devono stare molto attenti a quale proteina in particolare vorrebbero capire e investire quella quantità di denaro e di sforzi per poter trovare la struttura di quel gruppo.
Ora, se questo nuovo sistema permette ai ricercatori di farmaci di essere in grado non solo di capire la struttura 3D per un particolare target, ma anche un gran numero di target, questo apre sicuramente una finestra più grande per loro per capire non solo come un determinato farmaco interagirà con una proteina, ma anche come sarà tracciato con il modo in cui quel farmaco interagirà con molte altre proteine.
La nostra speranza è che questa conoscenza permetta agli esperimenti di diventare più efficaci. Ma questo è qualcosa che deve essere convalidato nel tempo.

Qual è il prossimo passo per voi ricercatori?
In questi ultimi tre o quattro anni ci siamo concentrati su questo problema estremamente importante e lo stiamo portando avanti. Siamo molto contenti di essere riusciti a fare questo passo avanti. Naturalmente ci sono ancora molti problemi, ed è su questo che si concentra il nostro team di ricerca: migliorare ulteriormente il sistema, non solo in termini di precisione, ma anche di copertura. Ci sono molte altre domande alle quali il nostro sistema non è ancora in grado di rispondere.
Per esempio, stiamo facendo previsioni sulla struttura statica di una proteina, ma le proteine non sono come i pezzi di Lego. Sono flessibili, quindi possono muoversi. Come si flettono e come questi movimenti permettono loro di legarsi ad altre proteine? Quando si pensa alla biologia, dove le proteine agiscono con altre proteine, come l’insulina, allora l’interazione diventa più sofisticata e la struttura flessibile deve essere presa in considerazione. Rispondere a queste domande è una sfida fondamentale per la scienza e la ricerca che intendiamo raccogliere in seguito.
Stiamo anche studiando il modo più impattante per far sì che questa tecnologia interagisca, non solo con il commercio, ma anche nelle partnership accademiche. Nelle prossime settimane il nostro team sta facendo dei piani su come fare delle partnership con partner accademici, industriali e commerciali. La cosa fondamentale che stiamo cercando di ottimizzare è l’impatto che questa tecnologia può avere nel mondo.